With 200 million predictions, what's the probability that my favorite protein won't make the list? 😭😭😭 #alphafoldwww.biorxiv.org/content/10.1...

Protein-protein interactions (PPI) are essential for biological function. Recent advances in coevolutionary analysis and Deep Learning (DL) based protein structure prediction have enabled comprehensiv...
😵 Computing the Human Interactome (200 million predicted protein structures) by the Baker lab #alphafoldwww.biorxiv.org/content/10.1...prodata.swmed.edu/humanPPI/

Protein-protein interactions (PPI) are essential for biological function. Recent advances in coevolutionary analysis and Deep Learning (DL) based protein structure prediction have enabled comprehensiv...
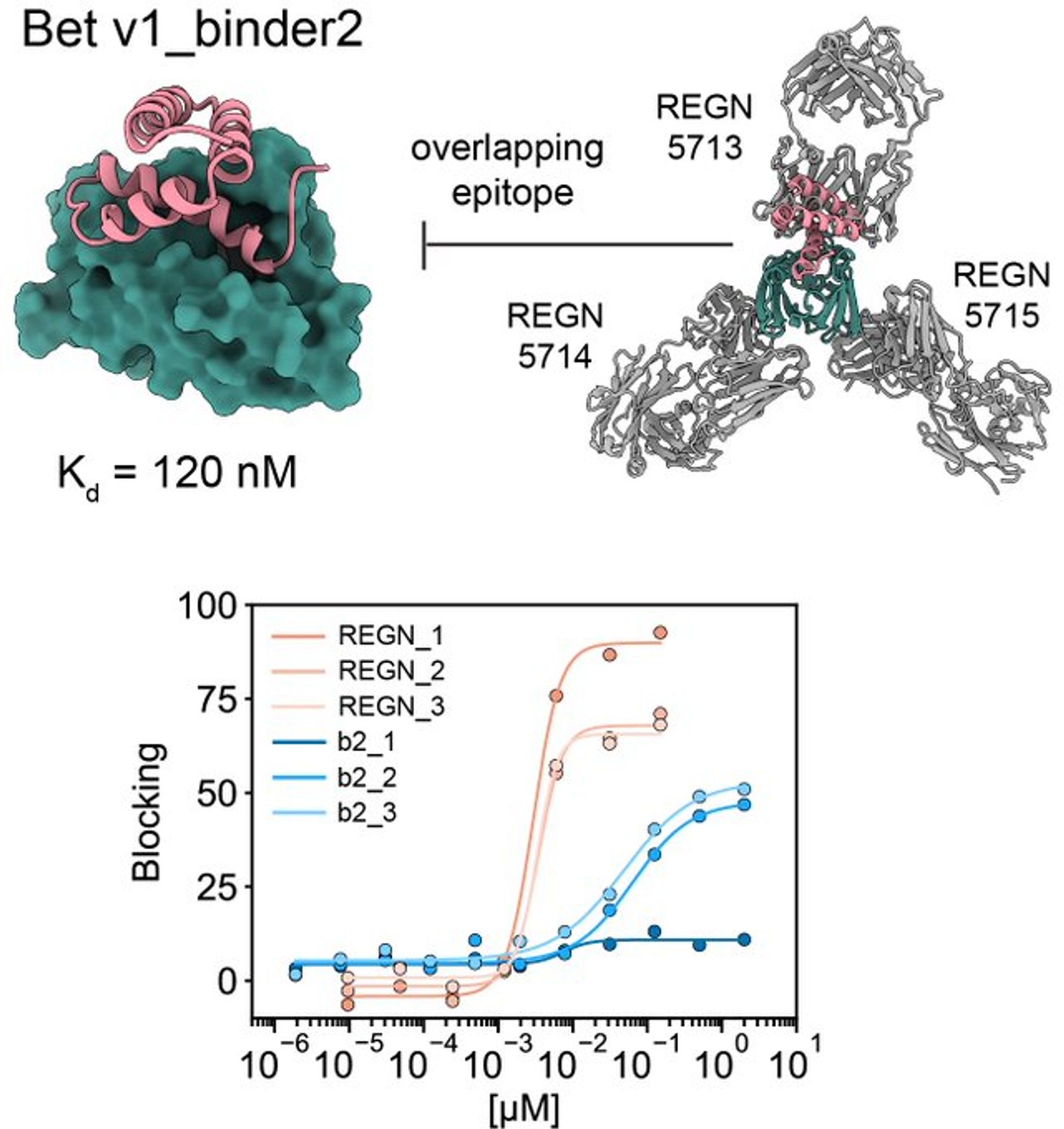
Cool preprint! AlphaFold has revolutionized protein structure prediction, but can it help design de novo protein binders too? This study takes a big step in that direction, with predictions and validations for binders targeting allergens, Cas9 and others. 🧪 www.biorxiv.org/content/10.1...
今後はAlphaFoldのタンパク質構造予測を筆頭に計算化学分野のAI創薬とかのテーマの受賞が増えそうだよね
Damit ist es der erste und einzige Ligand für diesen Rezeptor! (Und auch, welche Rezeptoren im Körper CXCL17 erkennen, ist bisher nicht ganz klar.) Um das AlphaFold-Modell zu bestätigen, haben sie einen live cell Assay durchgeführt, ⬇️
Die Forscher:innen haben hier AlphaFold genutzt, um die Bindung des Chemokins CXCL17 (Chemokine sind wichtige Botenstoffe des Immunsystems) an verschiedene Rezeptoren zu modellieren. Und tatsächlich kann laut AlphaFold der Orphan Rezeptor GPR25 das Chemokin CXCL17 binden. ⬇️
#GalaxyInResearchwww.nature.com/articles/s41...
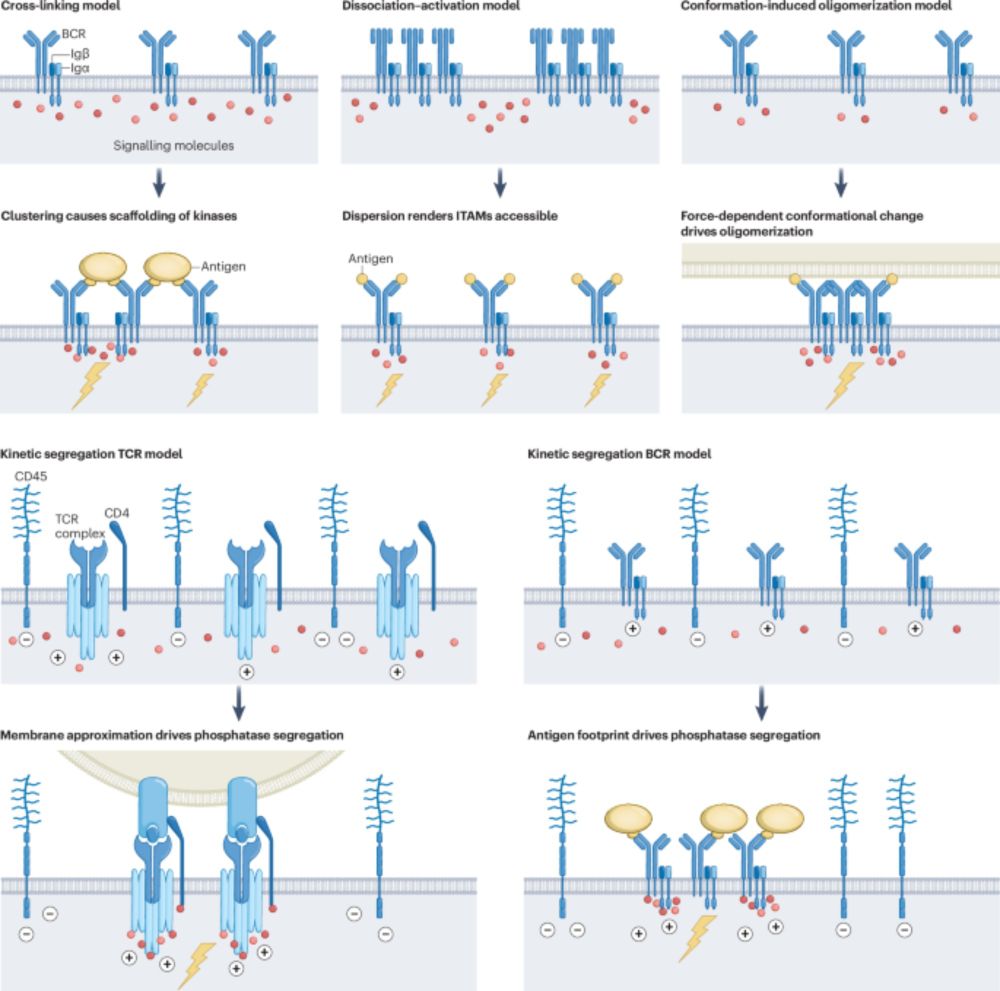
The mechanisms by which antigen triggers B-cell activation are incompletely understood. In this Review, Degn and Tolar discuss the different models of B-cell receptor triggering that have been propose...
Is there a self-help group for “I ran an AlphaFold 3 prediction and it is fundamentally different to the AF2 prediction”? Asking for a friend.
